今天被问到一个问题,基因结构注释中的 CDS1\CDS2\5U1E 是什么意思。简单来说是编码区,但是想想好像从未系统的梳理过基因结构注释结果中的各个概念。所以借此梳理以下。
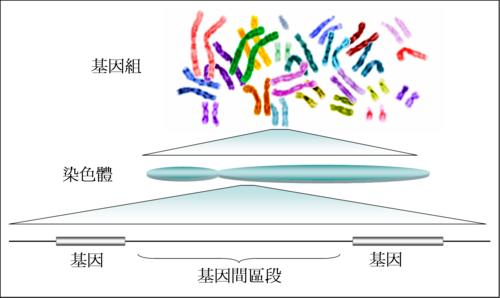
- 基因组
指一个生物体或细胞内所有遗传物质的总和。它不仅包含基因,还包含非基因序列(如端粒、着丝粒等)- DNA
即脱氧核糖核酸,是基因组的化学本质。- 核DNA
- 染色体
- 编码区: 直接参与蛋白质合成
- Exon: 基因中被转录并保留在成熟 mRNA 中的片段。
- 5U1E: 位于起始密码子之前。影响核糖体结合及翻译效率
- CDS : 严格指 mRNA 上从起始密码子到终止密码子之间、能被翻译成蛋白质的核苷酸序列。它对应于基因组中的外显子序列(除去 UTR 部分)
- 3U1E:位于终止密码子之后。包含 miRNA 结合位点,调控 mRNA 稳定性和翻译效率
- Intron:基因中位于两个外显子之间的间隔序列。在转录后被剪接体切除,不进入成熟 mRNA。
- Exon: 基因中被转录并保留在成熟 mRNA 中的片段。
- 非编码区: 不直接编码蛋白质,但负责调控和结构功能
- 启动子 (Promoter):通常位于基因转录起始位点的上游 (Upstream)(5’端附近);功能:RNA 聚合酶识别并结合的位置,决定转录是否开始
- 增强子 (Enhancer):可以位于基因的 上游、下游,甚至内含子中。无方向性。功能:像“加速器”一样显著增强启动子的活性,使基因在特定时空高表达。机制:通过染色质环化(Looping)物理接触启动子发挥作用。
- 编码区: 直接参与蛋白质合成
- 染色体
- 线粒体DNA
- 核DNA
- DNA
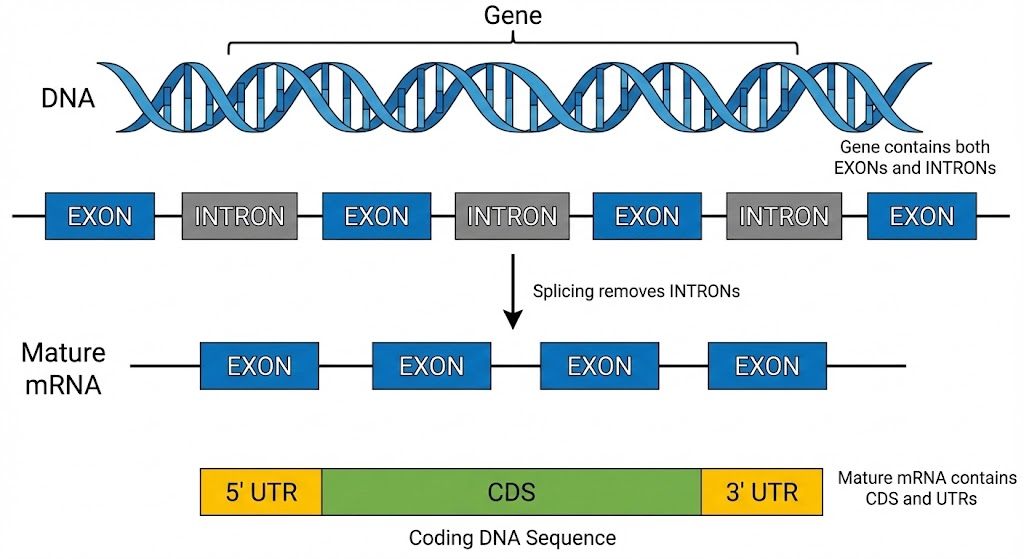
- 编码区(CDS)
- 以起始密码子ATG的第一个碱基A开始,并记为c.1,以终止密码子(TAA, TAG, TGA)的最后一个碱基为终点。
- 内含子区(Intron)
- 靠近内含子5’末端的变异位点,需依据上游最近外显子的最后一个碱基来定位,如c.87+4,代表上游最近外显子的边界位置为87,变异位点在内含子5’ 端开始的第4个碱基;
- 靠近内含子3’ 末端的变异位点,要依据下游最近外显子的第一个碱基来定位,如c.88-11,
- 内含子碱基个数为偶数时,中间碱基平分后按上下游外显子碱基来定位命名,如…,c.87+676, c.87+677, c.87+678, c.88-678, c.88-677, c.88-676, …
- 内含子碱基个数为奇数时,中间碱基相对于上游外显子最后一个碱基来定位命名,如…,c.87+677, c.87+678, c.87+679, c.88-678, c.88-677, …
- 非编码区(UTR区):
- 起始密码子ATG上游(5’ UTR区)标记为“-”,编号为c.-1, c.-2, c.-3…
- 终止密码子下游(3’ UTR区)标记为“”,编号为c.1, c.2, c.3…
- 位于靠近5’ UTR和3’ UTR区的内含子变异位点,命名规则同内含子区,如:5’ UTR区内含子为c.-85+1,c.-84-3等;3’ UTR区内含子为c.37+1,c.38-3等。
参考示意图如下: